随着高通量生物技术的不断发展,多种类型的生物组学数据大量产生,为彻底理解生命分子机理提供了前所未有的新契机。然而,如何有效集成分析这些组学数据破解生命秘密成为摆在生物学家、医学家以及信息科学家面前的一道难题。
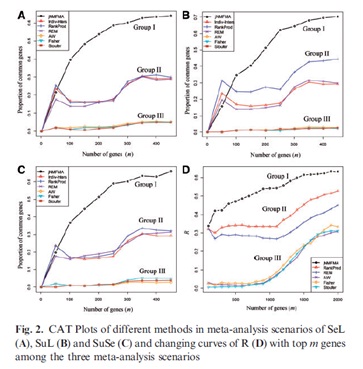
最近,合肥物质科学研究院智能机械研究所机器智能与计算生物学实验室王红强课题组开发出一种高通量数据meta分析方法。该方法巧妙地将联合矩阵分解技术与基因调控模型相结合,有效揭示了生命分子活动在不同遗传分子水平上的系统一致性,为深入探索生命活动规律提供了新的手段和途径。大量的模拟实验证实,该技术对肿瘤标记物的识别性能远远优于现有方法。研究结果也证实影响高通量meta分析的主要因素为数据依赖性和异质性。这两种因素将现有分析方法分为了三组,其性能从高到低具有显著的区别(如图)。结合非小细胞肺癌(NSCLC)的多组学数据,所提出的方法识别出了多种已知的甲基化驱动致癌基因,也发现了许多新的NSCLC标记物。这些新的标记物可用于开发新的NSCLC甲基化药物。
该成果发表在英国牛津大学出版集团的Bioinformatics 杂志最新一期上。在文章的评审过程中,两位审稿人都给了该工作以较高的评价:“方法的有用性和稳定性令人印象深刻”,“我认为这是一项非常有用的工作成果”。
该技术具有较强的通用性,在医学、农业以及细胞辐射机理研究都有广阔的应用前景。智能所机器智能与计算生物学实验室主要从事机器学习与智能算法及其在生物信息学方面的研究。近年来在生物信息学领域取得多项成果,主要发表在PLOS one、Bioinformatics 等国际杂志上。
文章链接
合肥研究院开发出基于高通量组学数据的肿瘤分子标记物识别方法

 联系我们
联系我们